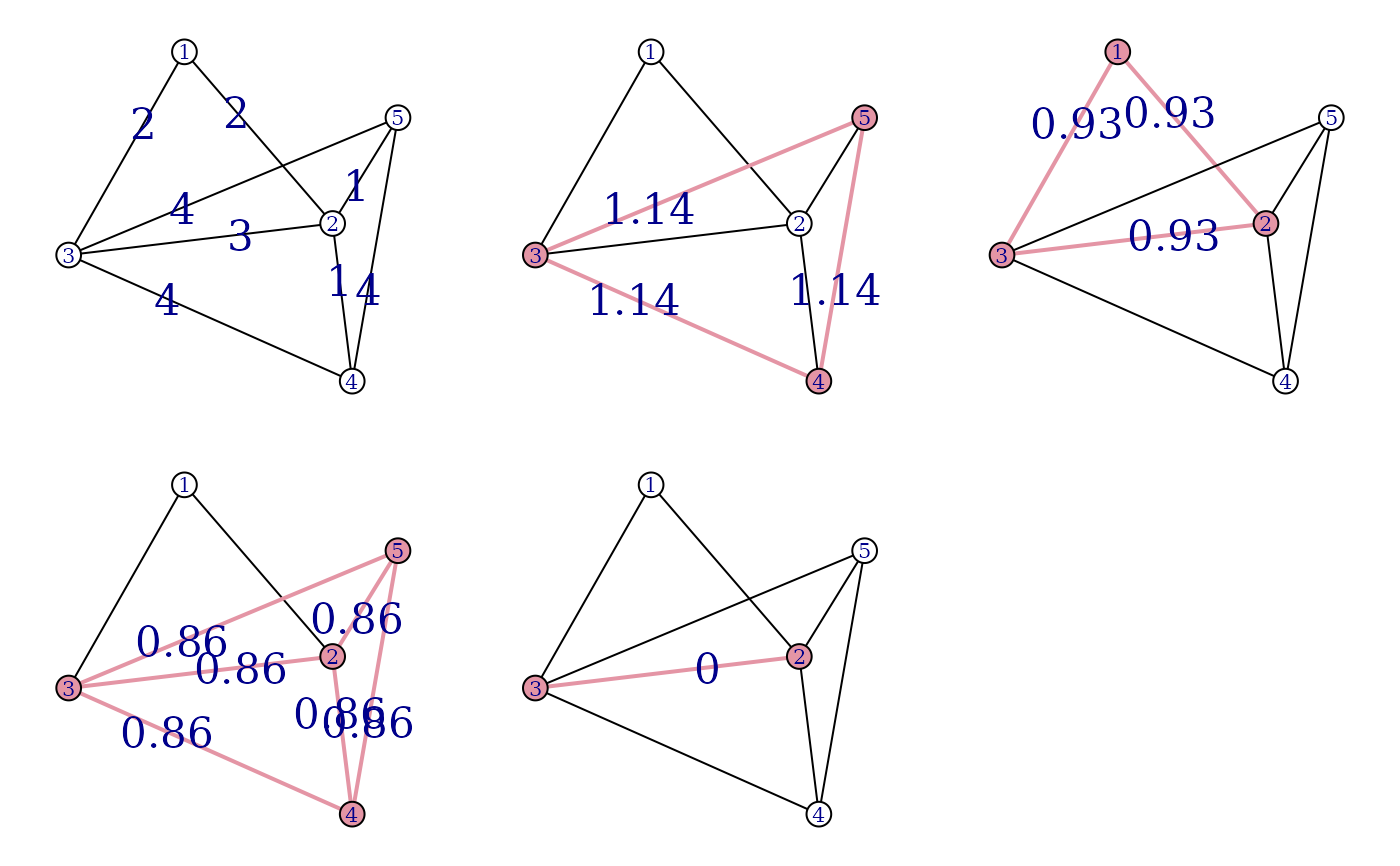
Graphlet 分解通过潜在重叠的密集社交组的联合来对加权无向图进行建模。这是通过一个两步算法完成的。第一步,通过找到阈值输入图中的团来创建一个候选组集(候选基)。第二步,将图投影到候选基上,从而为候选基中的每个团生成一个权重系数。
值
graphlets() 返回一个包含两个成员的列表
- cliques
子图列表,候选 graphlet 基。每个子图由顶点 ID 向量给出。
- Mu
graphlet 基中子图的权重。
graphlet_basis() 返回一个包含两个元素的列表
- cliques
子图列表,候选 graphlet 基。每个子图由顶点 ID 向量给出。
- thresholds
用于查找子图的权重阈值。
graphlet_proj() 返回一个数值向量,即 graphlet 基子图的权重。
详细信息
igraph 包含三个函数来执行图的分解。第一个是 graphlets(),它对该方法执行两个步骤,并返回一个子图列表及其相应的权重。第二个和第三个函数对应于该算法的第一步和第二步,如果用户希望单独执行它们,则它们很有用:graphlet_basis() 和 graphlet_proj()。
示例
## Create an example graph first
D1 <- matrix(0, 5, 5)
D2 <- matrix(0, 5, 5)
D3 <- matrix(0, 5, 5)
D1[1:3, 1:3] <- 2
D2[3:5, 3:5] <- 3
D3[2:5, 2:5] <- 1
g <- simplify(graph_from_adjacency_matrix(D1 + D2 + D3,
mode = "undirected", weighted = TRUE
))
V(g)$color <- "white"
E(g)$label <- E(g)$weight
E(g)$label.cex <- 2
E(g)$color <- "black"
layout(matrix(1:6, nrow = 2, byrow = TRUE))
co <- layout_with_kk(g)
par(mar = c(1, 1, 1, 1))
plot(g, layout = co)
## Calculate graphlets
gl <- graphlets(g, niter = 1000)
## Plot graphlets
for (i in 1:length(gl$cliques)) {
sel <- gl$cliques[[i]]
V(g)$color <- "white"
V(g)[sel]$color <- "#E495A5"
E(g)$width <- 1
E(g)[V(g)[sel] %--% V(g)[sel]]$width <- 2
E(g)$label <- ""
E(g)[width == 2]$label <- round(gl$Mu[i], 2)
E(g)$color <- "black"
E(g)[width == 2]$color <- "#E495A5"
plot(g, layout = co)
}