为 igraph 类的对象计算内聚块。
用法
cohesive_blocks(graph, labels = TRUE)
# S3 method for class 'cohesiveBlocks'
length(x)
blocks(blocks)
graphs_from_cohesive_blocks(blocks, graph)
# S3 method for class 'cohesiveBlocks'
cohesion(x, ...)
hierarchy(blocks)
parent(blocks)
# S3 method for class 'cohesiveBlocks'
print(x, ...)
# S3 method for class 'cohesiveBlocks'
summary(object, ...)
# S3 method for class 'cohesiveBlocks'
plot(
x,
y,
colbar = rainbow(max(cohesion(x)) + 1),
col = colbar[max_cohesion(x) + 1],
mark.groups = blocks(x)[-1],
...
)
plot_hierarchy(
blocks,
layout = layout_as_tree(hierarchy(blocks), root = 1),
...
)
export_pajek(blocks, graph, file, project.file = TRUE)
max_cohesion(blocks)参数
- graph
对于
cohesive_blocks(),是一个igraph类的图对象。它必须是无向的和简单的。(参见is_simple()。)对于
graphs_from_cohesive_blocks()和export_pajek(),必须提供相同的图,其内聚块结构在blocks()参数中给出。- labels
逻辑标量,是否将顶点标签添加到结果对象。这些标签可以在报告和绘制内聚块时使用。
- blocks, x, object
一个
cohesiveBlocks对象,使用cohesive_blocks()函数创建。- ...
附加参数。
plot_hierarchy()和plot()将它们传递给plot.igraph()。print()和summary()忽略它们。- y
图,其内聚块在
x参数中提供。- colbar
顶点颜色的颜色条。其长度应至少为 \(m+1\),其中 \(m\) 是图中最大的内聚度。或者,也可以通过
col参数直接指定顶点颜色。- col
顶点颜色的向量,以任何常用格式。(符号颜色名称(例如“red”、“blue”等),RGB 颜色(例如“#FF9900FF”),引用当前调色板的整数。默认情况下,使用给定的
colbar,并且具有相同最大内聚度的顶点将具有相同的颜色。- mark.groups
要在图中标记的顶点集合列表,通过用圆圈圈起来。默认情况下,标记所有内聚块,除了对应于所有顶点的那个。
- layout
绘图的布局,它只是传递给
plot.igraph(),参见那里的可能格式。默认情况下,使用 Reingold-Tilford 布局生成器。- file
定义 Pajek 文件写入的文件(或连接)。
如果
project.file参数为TRUE,则它可以是文件名(带扩展名)、文件对象,或者通常是任何类型的连接对象。如果文件/连接尚未打开,则会将其打开。如果
project.file参数为FALSE,则会创建多个文件,并且file必须是一个字符标量,其中包含文件的基本名称,不带扩展名。(但它可以包含文件的路径。)另请参见下面的详细信息。
- project.file
逻辑标量,是否创建包含所有数据的单个 Pajek 项目文件,或者为每个项目创建单独的文件。请参阅下面的详细信息。
值
cohesive_blocks() 返回一个 cohesiveBlocks 对象。
blocks() 返回一个数字向量列表,其中包含顶点 ID。
graphs_from_cohesive_blocks() 返回一个 igraph 图列表,对应于内聚块。
cohesion() 返回一个数值向量,即每个块的内聚度。
hierarchy() 返回一个 igraph 图,表示内聚块的层次结构。
parent() 返回一个数值向量,给出块层次结构中每个内聚块的父块。层次结构根部的块没有父块,并为其返回 0。
plot_hierarchy()、plot() 和 export_pajek() 返回 NULL,不可见。
max_cohesion() 返回一个数值向量,每个顶点一个条目,给出其最内聚块的内聚度。
print() 和 summary() 返回 cohesiveBlocks 对象本身,不可见。
length 返回一个数值标量,即块的数量。
详细信息
内聚块是一种基于图顶点的结构内聚性(或顶点连通性)确定图顶点层次子集的方法。对于给定的图 \(G\),如果 \(S\) 没有顶点连通性大于或等于 \(k\) 的超集,则其顶点子集 \(S\subset V(G)\) 被称为最大 \(k\) 内聚。内聚块是一个过程,通过该过程,给定一个 \(k\) 内聚顶点集,递归地识别最大 \(l\) 内聚子集,其中 \(l>k\)。因此,找到一个顶点子集的层次结构,整个图 \(G\) 位于其根部。
函数 cohesive_blocks() 实现内聚块。它返回一个 cohesiveBlocks 对象。cohesiveBlocks 应被视为不透明类,即不应直接访问其内部结构,而是通过此处列出的函数。
函数 length 可用于 cohesiveBlocks 对象,它给出块的数量。
函数 blocks() 返回存储在 cohesiveBlocks 对象中的实际块。它们以数字向量列表的形式返回,每个向量包含顶点 ID。
函数 graphs_from_cohesive_blocks() 类似,但将块作为输入图的(导出)子图返回。各种(图、顶点和边)属性都保存在子图中。
函数 cohesion() 返回一个数字向量,即不同块的内聚度。块的顺序与 blocks() 和 graphs_from_cohesive_blocks() 函数相同。
可以使用 hierarchy() 函数查询块层次结构。它返回一个 igraph 图,其顶点 ID 根据 blocks() 和 graphs_from_cohesive_blocks()、cohesion() 等函数中块的顺序排序。
parent() 给出每个块在其块层次结构中的父顶点,对于根顶点,它给出 0。
plot_hierarchy() 通过调用 igraph.plot 在活动的图形设备上绘制内聚块的层次结构树。
export_pajek() 函数可用于以 Pajek 格式导出图及其内聚块。它可以导出包含所有信息的单个 Pajek 项目文件,也可以导出一组文件,具体取决于其 project.file 参数。如果 project.file 为 TRUE,则以下信息将写入 file 参数中给出的文件(或连接):(1) 输入图及其属性,有关详细信息,请参阅 write_graph();(2) 层次结构图;(3) 每个内聚块一个二元分区。如果 project.file 为 FALSE,则 file 参数必须是字符标量,并用作生成文件的基本名称。如果 file 为“basename”,则会创建以下文件:(1) 原始图的“basename.net”;(2) 层次结构图的“basename_hierarchy.net”;(3) 每个内聚块的“basename_block_x.net”,其中“x”是块的编号,从一开始。
max_cohesion() 返回每个顶点的最大内聚度,即顶点最内聚块的内聚度。
泛型函数 summary() 可用于 cohesiveBlocks 对象,它会将一行摘要打印到终端。
泛型函数 print() 也定义在 cohesiveBlocks 对象上,如果在键入 cohesiveBlocks 对象的名称时会自动调用它。它会生成如下输出
Cohesive block structure:
B-1 c 1, n 23
'- B-2 c 2, n 14 oooooooo.. .o......oo ooo
'- B-4 c 5, n 7 ooooooo... .......... ...
'- B-3 c 2, n 10 ......o.oo o.oooooo.. ...
'- B-5 c 3, n 4 ......o.oo o......... ... 左侧部分显示块结构,在本例中为五个块。第一个块始终对应于整个图,即使其内聚度为零。然后显示块的内聚度和块中顶点的数量。仅当显示足够宽时才会打印最后一部分,并按顶点 ID 显示块中的顶点。“o”表示包含顶点,点表示不包含顶点,并且顶点以十个为一组显示。
泛型函数 plot() 绘制图,显示其中的一个或多个内聚块。
参考文献
J. Moody 和 D. R. White。Structural cohesion and embeddedness: A hierarchical concept of social groups。American Sociological Review, 68(1):103–127, Feb 2003, doi:10.2307/3088904 。
作者
Gabor Csardi csardi.gabor@gmail.com 用于当前实现,Peter McMahan (https://socialsciences.uchicago.edu/news/alumni-profile-peter-mcmahan-phd17-sociology) 在 R 中编写了第一个版本。
示例
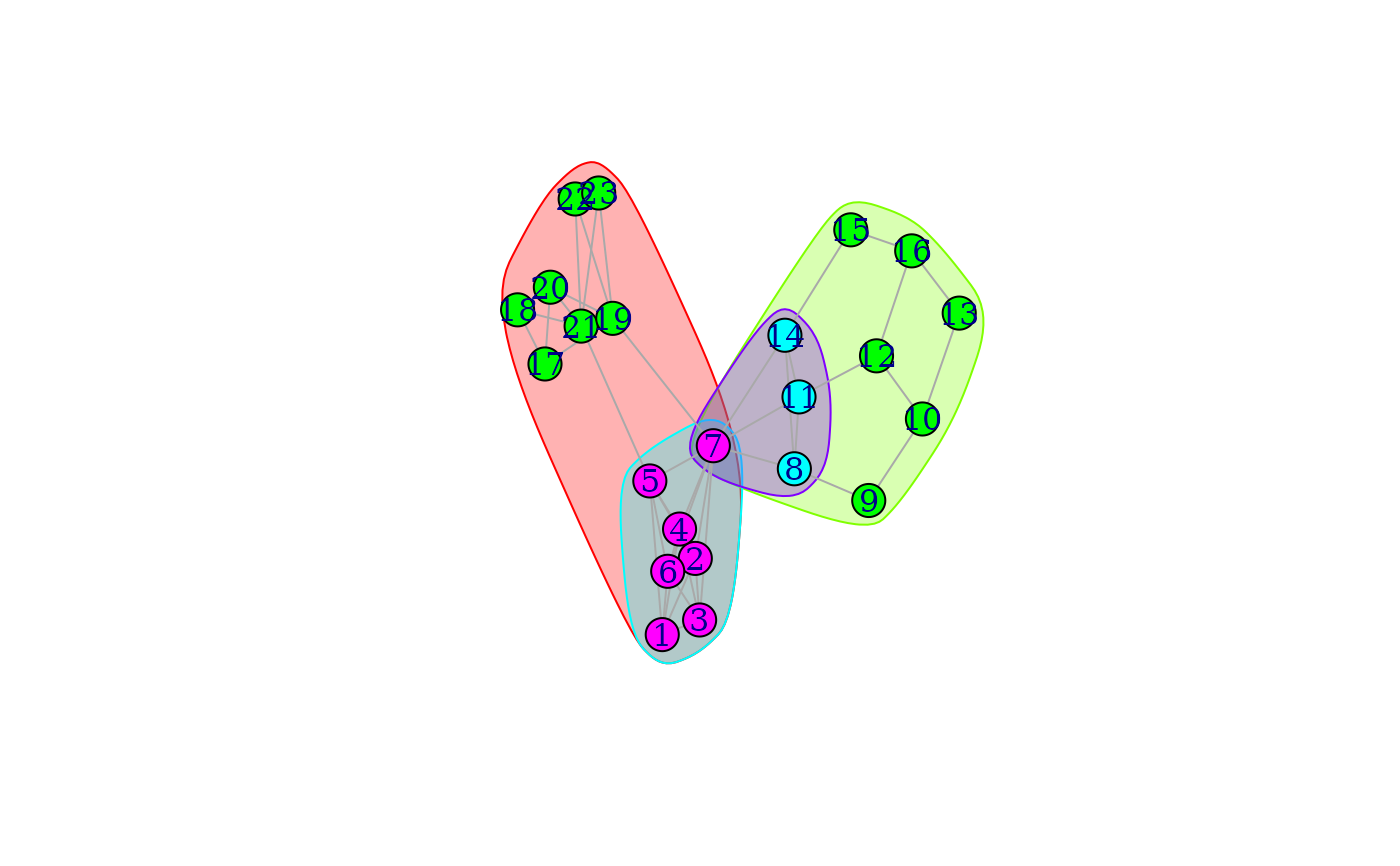
## The graph from the Moody-White paper
mw <- graph_from_literal(
1 - 2:3:4:5:6, 2 - 3:4:5:7, 3 - 4:6:7, 4 - 5:6:7,
5 - 6:7:21, 6 - 7, 7 - 8:11:14:19, 8 - 9:11:14, 9 - 10,
10 - 12:13, 11 - 12:14, 12 - 16, 13 - 16, 14 - 15, 15 - 16,
17 - 18:19:20, 18 - 20:21, 19 - 20:22:23, 20 - 21,
21 - 22:23, 22 - 23
)
mwBlocks <- cohesive_blocks(mw)
# Inspect block membership and cohesion
mwBlocks
#> Cohesive block structure:
#> B-1 c 1, n 23
#> '- B-2 c 2, n 14 oooooooo.. .o......oo ooo
#> '- B-4 c 5, n 7 ooooooo... .......... ...
#> '- B-3 c 2, n 10 ......o.oo o.oooooo.. ...
#> '- B-5 c 3, n 4 ......o.oo o......... ...
blocks(mwBlocks)
#> [[1]]
#> + 23/23 vertices, named, from b122831:
#> [1] 1 2 3 4 5 6 7 21 8 11 14 19 9 10 12 13 16 15 17 18 20 22 23
#>
#> [[2]]
#> + 14/23 vertices, named, from b122831:
#> [1] 1 2 3 4 5 6 7 21 19 17 18 20 22 23
#>
#> [[3]]
#> + 10/23 vertices, named, from b122831:
#> [1] 7 8 11 14 9 10 12 13 16 15
#>
#> [[4]]
#> + 7/23 vertices, named, from b122831:
#> [1] 1 2 3 4 5 6 7
#>
#> [[5]]
#> + 4/23 vertices, named, from b122831:
#> [1] 7 8 11 14
#>
cohesion(mwBlocks)
#> [1] 1 2 2 5 3
# Save results in a Pajek file
file <- tempfile(fileext = ".paj")
export_pajek(mwBlocks, mw, file = file)
if (!interactive()) {
unlink(file)
}
# Plot the results
plot(mwBlocks, mw)
 ## The science camp network
camp <- graph_from_literal(
Harry:Steve:Don:Bert - Harry:Steve:Don:Bert,
Pam:Brazey:Carol:Pat - Pam:Brazey:Carol:Pat,
Holly - Carol:Pat:Pam:Jennie:Bill,
Bill - Pauline:Michael:Lee:Holly,
Pauline - Bill:Jennie:Ann,
Jennie - Holly:Michael:Lee:Ann:Pauline,
Michael - Bill:Jennie:Ann:Lee:John,
Ann - Michael:Jennie:Pauline,
Lee - Michael:Bill:Jennie,
Gery - Pat:Steve:Russ:John,
Russ - Steve:Bert:Gery:John,
John - Gery:Russ:Michael
)
campBlocks <- cohesive_blocks(camp)
campBlocks
#> Cohesive block structure:
#> B-1 c 2, n 18
#> '- B-2 c 3, n 4 oooo...... ........
#> '- B-3 c 3, n 5 ....ooooo. ........
#> '- B-4 c 3, n 6 .........o ooooo...
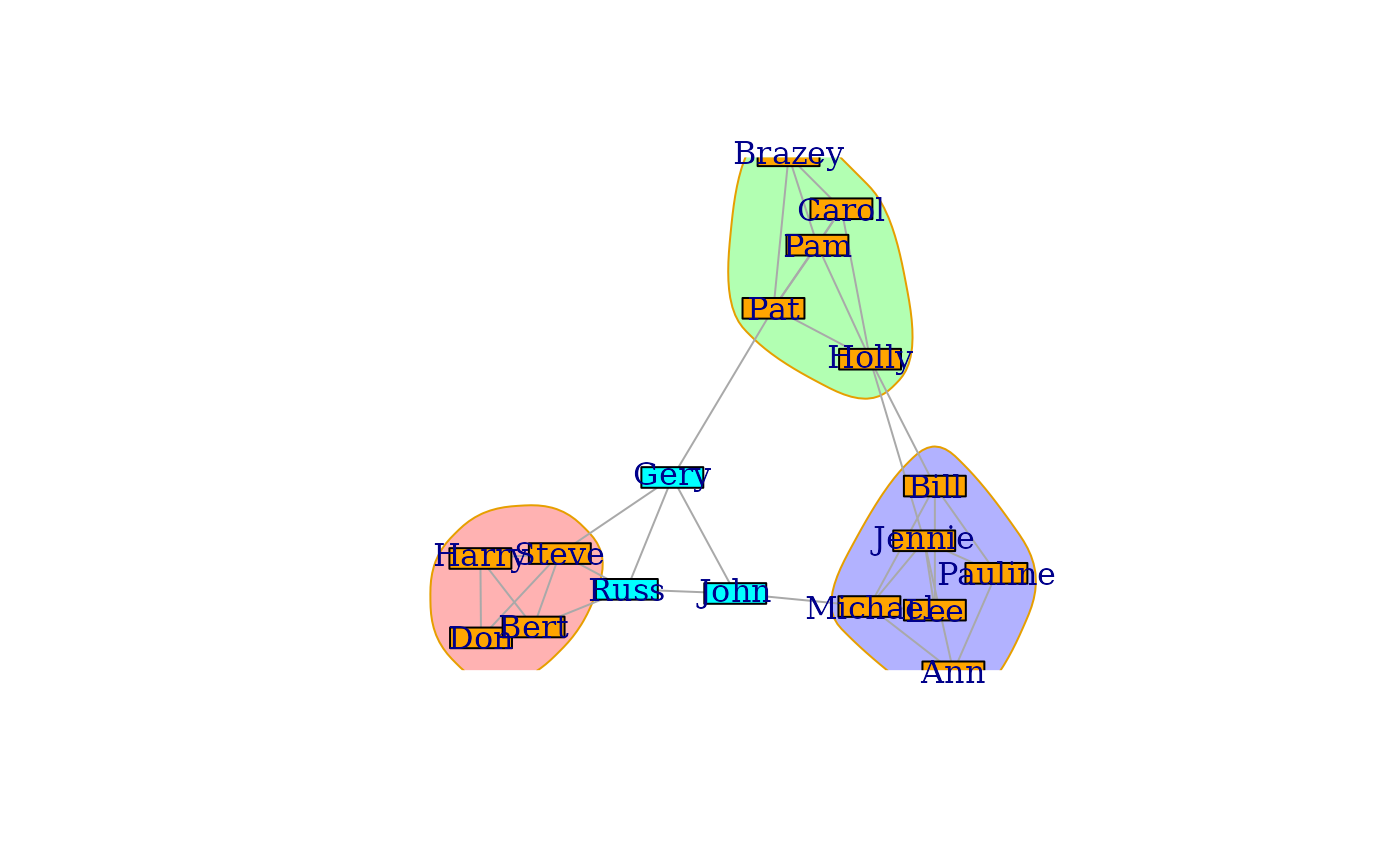
plot(campBlocks, camp,
vertex.label = V(camp)$name, margin = -0.2,
vertex.shape = "rectangle", vertex.size = 24, vertex.size2 = 8,
mark.border = 1, colbar = c(NA, NA, "cyan", "orange")
)
## The science camp network
camp <- graph_from_literal(
Harry:Steve:Don:Bert - Harry:Steve:Don:Bert,
Pam:Brazey:Carol:Pat - Pam:Brazey:Carol:Pat,
Holly - Carol:Pat:Pam:Jennie:Bill,
Bill - Pauline:Michael:Lee:Holly,
Pauline - Bill:Jennie:Ann,
Jennie - Holly:Michael:Lee:Ann:Pauline,
Michael - Bill:Jennie:Ann:Lee:John,
Ann - Michael:Jennie:Pauline,
Lee - Michael:Bill:Jennie,
Gery - Pat:Steve:Russ:John,
Russ - Steve:Bert:Gery:John,
John - Gery:Russ:Michael
)
campBlocks <- cohesive_blocks(camp)
campBlocks
#> Cohesive block structure:
#> B-1 c 2, n 18
#> '- B-2 c 3, n 4 oooo...... ........
#> '- B-3 c 3, n 5 ....ooooo. ........
#> '- B-4 c 3, n 6 .........o ooooo...
plot(campBlocks, camp,
vertex.label = V(camp)$name, margin = -0.2,
vertex.shape = "rectangle", vertex.size = 24, vertex.size2 = 8,
mark.border = 1, colbar = c(NA, NA, "cyan", "orange")
)