igraph 社群检测函数将其结果作为 communities 类的对象返回。此手册页描述了此类的操作。
用法
membership(communities)
# S3 method for class 'communities'
print(x, ...)
# S3 method for class 'communities'
modularity(x, ...)
# S3 method for class 'communities'
length(x)
sizes(communities)
algorithm(communities)
merges(communities)
crossing(communities, graph)
code_len(communities)
is_hierarchical(communities)
# S3 method for class 'communities'
as.dendrogram(object, hang = -1, use.modularity = FALSE, ...)
# S3 method for class 'communities'
as.hclust(x, hang = -1, use.modularity = FALSE, ...)
cut_at(communities, no, steps)
show_trace(communities)
# S3 method for class 'communities'
plot(
x,
y,
col = membership(x),
mark.groups = communities(x),
edge.color = c("black", "red")[crossing(x, y) + 1],
...
)
communities(x)参数
- communities, x, object
一个
communities对象,igraph 社群检测函数的结果。- ...
附加参数。
plot.communities将这些参数传递给plot.igraph()。其他函数会静默地忽略它们。- graph
一个 igraph 图对象,对应于
communities。- hang
数值标量,指示应如何根据其父节点的高度计算叶节点的高度;请参见
plot.hclust()。- use.modularity
逻辑标量,指示是否使用模块化值来定义分支的高度。
- no
整数标量,表示所需的社群数量。如果太低或太高,则会给出错误消息。必须提供
no和steps中的一个。- steps
要执行的合并操作数以生成社群。必须提供
no和steps中的一个。- y
一个 igraph 图对象,对应于
x中的社群。- col
一个颜色向量,格式为常规 R 绘图方法接受的任何格式。此向量显式地给出了顶点的颜色。
- mark.groups
一个数值向量列表。可以使用彩色多边形突出显示社群。此处给出了绘制多边形的组。默认是使用社群给出的组。如果不想突出显示任何组,请在此处提供
NULL。- edge.color
边的颜色。默认情况下,社群内的边为绿色,其他边为红色。
- membership
数值向量,每个顶点一个值,社群结构的成员向量。如果社群结构以另一种方式给出,例如通过合并矩阵,则也可能为
NULL。- algorithm
如果不是
NULL(表示未知算法),则为字符标量,即生成社群结构的算法的名称。- merges
如果不是
NULL,则为分层社群结构的合并矩阵。有关其格式的更多信息,请参见下面的merges()。- modularity
数值标量或向量,社群结构的模块化值。如果(最佳)分割的模块化不可用,则也可以为
NULL。
值
print() 以不可见的方式返回 communities 对象本身。
length 返回一个整数标量。
sizes() 返回一个数值向量。
membership() 返回一个数值向量,该图中的每个顶点一个数字,该顶点是社群检测的输入。
modularity() 返回一个数值标量。
algorithm() 返回一个字符标量。
crossing() 返回一个逻辑向量。
is_hierarchical() 返回一个逻辑标量。
merges() 返回一个两列的数值矩阵。
cut_at() 返回一个数值向量,即顶点的成员向量。
as.dendrogram() 返回一个 dendrogram 对象。
show_trace() 返回一个字符向量。
code_len() 为使用 InfoMAP 方法找到的社群返回一个数值标量,为其他方法返回 NULL。
plot() 对于 communities 对象返回 NULL,不可见。
详细信息
社群结构检测算法尝试通过优化某些标准,通常使用启发法来找到有向或无向图中的密集子图。
igraph 实现了许多社群检测方法(请参见下面的内容),所有这些方法都返回一个 communities 类的对象。由于社群结构检测算法不同,因此 communities 对象并不总是具有相同的结构。但是,它们有一些共同的操作,这些操作在此处进行了说明。
为 communities 定义了 print() 泛型函数,它会打印一个简短的摘要。
可以在 communities 上调用 length 泛型函数,并返回社群数。
sizes() 函数以其 ID 的顺序返回社群大小。
membership() 给出了顶点到社群中的划分。它返回一个数值向量,每个顶点一个值,即其社群的 ID。社群 ID 从 1 开始。请注意,某些算法会计算社群的完整(或不完整)的分层结构,而不仅仅是单个分区。对于这些算法,通常会返回最高模块化值的成员资格,但也请参见各个算法的手册页。
communities() 也是一个函数的名称,该函数返回社群列表,每个社群都由其顶点标识。如果设置了 add.vertex.names igraph 选项,并且图本身已命名,则顶点将具有符号名称。否则,将使用数字顶点 ID。
modularity() 给出了分区的模块化分数。(有关详细信息,请参见 modularity.igraph()。对于不产生单个分区的算法,将返回最高模块化值。
algorithm() 给出了用于计算社群结构的算法的名称。
crossing() 返回一个逻辑向量,每个边一个值,按边 ID 排序。如果该边连接两个不同的社群,则该值为 TRUE,根据 membership() 返回的(最佳)成员向量。
is_hierarchical() 检查是否使用了分层算法来查找社群结构。某些函数仅对分层方法有意义(例如 merges()、cut_at() 和 as.dendrogram())。
merges() 返回分层方法的合并矩阵。如果使用非分层方法来查找社群结构,则会给出错误消息。您可以通过在 communities 对象上调用 is_hierarchical() 来检查这一点。
cut_at() 在所需的位置剪切分层社群查找方法的合并树,并返回成员向量。所需的位置可以表示为所需的社群数量或要进行的合并步骤数。如果使用非分层方法调用该函数,则会给出错误消息。
as.dendrogram() 将分层社群结构转换为 dendrogram 对象。它仅适用于分层方法,并向其他方法给出错误消息。有关详细信息,请参见 stats::dendrogram()。
stats::as.hclust() 类似于 as.dendrogram(),但将分层社群结构转换为 hclust 对象。
ape::as.phylo() 将分层社群结构转换为 phylo 对象,您需要 ape 包才能执行此操作。
show_trace()(当前)仅适用于通过前导特征向量方法找到的社群 (cluster_leading_eigen()),并返回一个字符向量,其中给出了算法在查找社群时执行的步骤。
为 InfoMAP 方法定义了 code_len() (cluster_infomap() 并返回分区的代码长度。
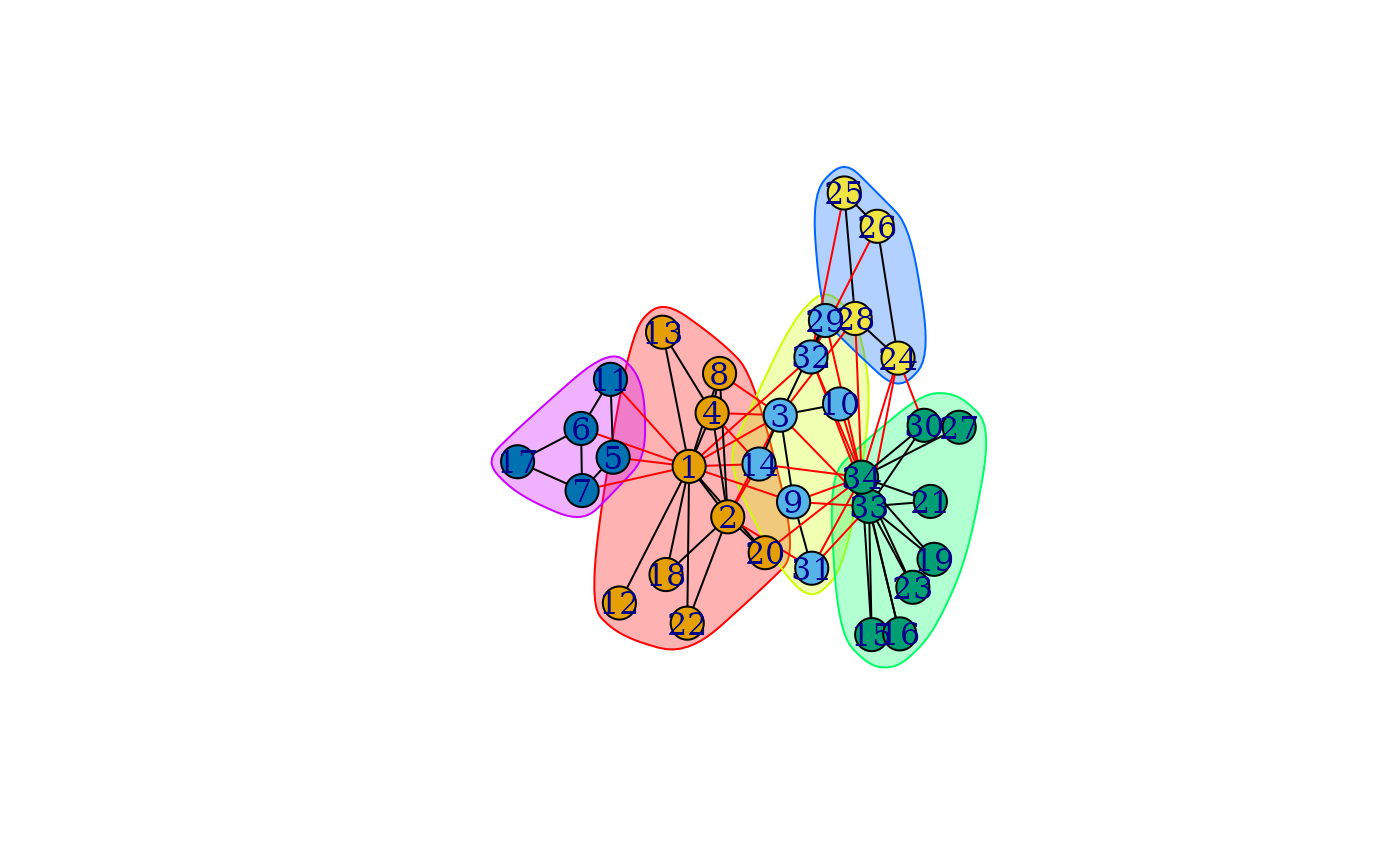
可以在 communities 对象上调用 plot() 函数。这将绘制图形(并在内部使用 plot.igraph()),并显示社群。默认情况下,它会根据其社群对顶点进行着色,并标记与社群相对应的顶点组。它将附加参数传递给 plot.igraph(),请参见该参数以及 igraph.plotting,了解如何更改绘图。
参见
有关绘制社群结构树状图的信息,请参见 plot_dendrogram()。
有关比较同一图上的两个社群结构的信息,请参见 compare()。
社群检测 as_membership()、cluster_edge_betweenness()、cluster_fast_greedy()、cluster_fluid_communities()、cluster_infomap()、cluster_label_prop()、cluster_leading_eigen()、cluster_leiden()、cluster_louvain()、cluster_optimal()、cluster_spinglass()、cluster_walktrap()、compare()、groups()、make_clusters()、modularity.igraph()、plot_dendrogram()、split_join_distance()、voronoi_cells()
作者
Gabor Csardi csardi.gabor@gmail.com
示例
karate <- make_graph("Zachary")
wc <- cluster_walktrap(karate)
modularity(wc)
#> [1] 0.3532216
membership(wc)
#> [1] 1 1 2 1 5 5 5 1 2 2 5 1 1 2 3 3 5 1 3 1 3 1 3 4 4 4 3 4 2 3 2 2 3 3
plot(wc, karate)